Labtest lança teste de biologia molecular para COVID-19
10 set 2020
A Labtest lança em seu portfólio um produto específico para o diagnóstico molecular do novo coronavírus, SARS-CoV-2, que causa a doença denominada pela OMS de COVID-19.1
O SARS-CoV-2, coronavírus da síndrome respiratória aguda severa 2, é um betacoronavírus de RNA fita simples positivo (+ssRNA). É um vírus envelopado que possui quatro proteínas estruturais: S (spike), E (envelope), M (membrana) e N (nucleocapsídeo). A proteína S é a responsável pela ligação do vírus à célula hospedeira permitindo a incorporação do material genético viral, viabilizando o processo de replicação do vírus.
Embora estudos recentes sugiram que o tempo médio de incubação do vírus seja de 5 dias, este período, que compreende o momento da infecção até o aparecimento de sintomas, pode variar.2
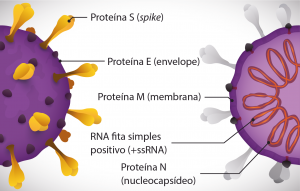
Figura 1. Representação esquemática da estrutura do vírus SARS-CoV-2.
O produto Detect SARS-CoV-2 (RT-PCR) Ref. 804 emprega a metodologia de RT-PCR em tempo real (reação em cadeia da polimerase em tempo real precedida de transcrição reversa). Considerada atualmente, a metodologia “padrão-ouro” para diagnóstico da COVID-19, este é o método de escolha para diagnóstico nos estágios iniciais da doença, conforme mostrado na Figura 2.
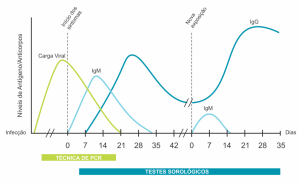
Figura 2. Resposta imunológica após a infecção pelo novo coronavírus (SARS-CoV-2).
PCR em Tempo Real: Aplicações e Protocolos para SARS-CoV-2
A reação em cadeia da polimerase em tempo real (PCR em tempo real) foi descrita em 1992 por Higuchi e colaboradores³ e é empregada na detecção e quantificação de ácidos nucleicos presentes no material genético de diferentes organismos. Na PCR em tempo real, os processos de amplificação e detecção ocorrem de maneira simultânea, diferente da PCR convencional, em que estes processos ocorrem de forma separada. Entre as vantagens da técnica de PCR em tempo real destacam-se: alta sensibilidade, boa reprodutibilidade e menor probabilidade de contaminação, pois não precisa de pós-processamento.
Se o material a ser avaliado for constituído de DNA, a PCR pode ser utilizada diretamente. Contudo, se o material a ser avaliado for constituído de RNA, como no caso do material extraído de amostras de pacientes infectados com SARS-CoV-2, a reação em cadeia da polimerase deve ser precedida de transcrição reversa, RT-PCR.
A transcrição reversa é o processo no qual o ácido ribonucleico (RNA) é convertido em ácido desoxirribonucleico complementar (cDNA). Desta maneira, a primeira etapa que ocorre na técnica de RT-PCR é a conversão do RNA extraído da amostra, previamente selecionada, em cDNA. A partir da presença do cDNA, decorrem simultaneamente os processos de amplificação e detecção do material genético amplificado.
A escolha dos genes a serem amplificados é uma etapa fundamental para garantir a especificidade do protocolo. Esta escolha deve ser baseada no sequenciamento genético do organismo a ser identificado.
O processo de detecção é feito por meio de sondas que contêm fluoróforos. Os fluoróforos são moléculas capazes de emitir um sinal fluorescente, permitindo a detecção do produto de PCR – cópias do gene que foi amplificado de maneira exponencial.
Entre as sondas mais utilizadas está a TaqManTM, uma sonda de hidrólise que contém oligonucleotídeos ligados a um fluoróforo (repórter) na extremidade 5’ e a um quencher na extremidade 3’. A função do quencher é absorver a fluorescência do repórter, enquanto estes estiverem ligados ao oligonucleotídeo. Esse tipo de sonda se liga de forma específica ao produto de PCR e, quando a polimerização encontra a sonda, ocorre a clivagem da mesma, pela ação da enzima Taq polimerase. O resultado da clivagem da sonda é a liberação do fluoróforo e consequente emissão de luz, detectada em um comprimento de onda específico.
A cada ciclo de PCR em tempo real ocorrem as etapas de desnaturação, anelação e extensão. Na desnaturação ocorre o rompimento das ligações de hidrogênio na fita de DNA com perda de sua estrutura tridimensional. Durante a anelação tem-se o reconhecimento dos iniciadores (primers) e sondas ao segmento do gene a ser amplificado e detectado. Já na extensão ocorre a polimerização da nova fita de DNA, no sentido 5’-3’, com a obtenção do produto de PCR e detecção deste por fluorescência. Este processo é mostrado na Figura 3.
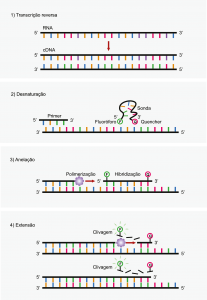
Figura 3. Sequência de processos de um ciclo de PCR em Tempo Real – Método TaqManTM.
Os protocolos para identificação do SARS-CoV-2 foram desenvolvidos logo após o sequenciamento genético do vírus. Os protocolos aprovados pela WHO (World Health Organization) e pelo CDC (Central for Disease Control) têm sido utilizados em diversas partes do mundo.
No protocolo do CDC são identificadas duas regiões diferentes do gene N, denominadas N1 e N2, e este utiliza como controle interno o gene RP (RNAse P humano). O fluoróforo empregado para detecção é o FAMTM.⁴ Já o protocolo adotado pela WHO, também conhecido como protocolo de Charité ou Europeu, identifica os genes RdRp, E e N. Este recomenda o gene E como triagem e o gene RdRp como confirmatório. O protocolo Charité foi avaliado em diferentes vírus e bactérias causadores de doenças respiratórias como: influenza, parainfluenza e outros coronavírus humanos, não sendo observadas reações cruzadas.⁵
Apesar de ser considerada metodologia “padrão-ouro” para diagnóstico da COVID-19, a técnica de RT-PCR apresenta algumas limitações: o material genético do vírus (RNA) é muito lábil e se degrada facilmente, portanto, a coleta e armazenamento da amostra devem ser realizados de forma adequada para minimizar os erros na fase pré-analítica. Também é importante evitar a contaminação dos materiais utilizados no ensaio com RNAses (enzimas capazes de degradar o RNA). Ademais, cabe ressaltar que a sensibilidade da RT-PCR é dependente do tipo de amostra, conforme mostrado por Wang e colaboradores⁶ (Figura 4).
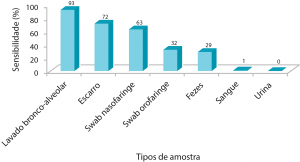
Figura 4. Gráfico da sensibilidade da técnica de RT-PCR para SAR-CoV-2 em função do tipo de amostra
Solução Labtest para detecção do SARS-CoV-2 por PCR em Tempo Real
O produto Detect SARS-CoV-2 (RT-PCR) Ref. 804 identifica três genes do SARS-CoV-2: RdRp, E e N. O sistema conta com sondas específicas, tipo TaqManTM, marcadas com os fluoróforos FAM™, Texas Red™ e JOE™, e uma sonda marcada com o fluoróforo Cy5™, para controle interno. O produto pode ser utilizado com RNA extraído de amostras do trato respiratório inferior (lavado bronco-alveolar e escarro humano) e do trato respiratório superior (swab de nasofaringe e swab de orofaringe).
O sistema é composto de: Mix de Reação, Mix de Sonda, Controle Positivo e Controle Negativo. Para cada reação são necessários 5 µL de RNA e 15 µL de Master Mix, que é preparado a partir do Mix de Reação e do Mix de Sonda. Os resultados podem ser obtidos em cerca de 120 minutos e a amostra é considerada positiva se forem identificados, pelo menos, um dos seguintes genes: RdRp ou N.
O comparativo foi realizado com RNA extraído de 120 amostras, sendo 60 positivas e 60 negativas, e o produto apresentou sensibilidade e especificidade de 100%.
Nenhuma reatividade cruzada foi observada com os seguintes agentes etiológicos: vírus influenza A (H1N1/09, H3N2 e H5N1), vírus influenza B, rinovírus, vírus sincicial respiratório (A/B), vírus da parainfluenza 1, vírus da parainfluenza 2, vírus da parainfluenza 3, vírus da parainfluenza 4, adenovírus, bocavírus humano, sarampo e Mycoplasma spp.

Figura 5. Imagem ilustrativa do produto Detect SARS-CoV-2 (RT-PCR) Ref. 804.
O reagente Detect SARS-CoV-2 (RT-PCR) Ref. 804 está disponível na apresentação de 100 determinações.
Produto de uso profissional, somente para diagnóstico in vitro.
Conte com a excelência técnico-científica da Labtest
Além de oferecer soluções de qualidade e confiança, os produtos com a marca Labtest têm o compromisso maior com a VIDA. A reconhecida excelência da Assessoria Técnico-Científica da Labtest oferece todo o suporte necessário para o acompanhamento do desempenho dos reagentes e suas aplicações, seja no DDG 0800 031 3411 ou no sac@labtest.combr
Para novas informações, entre em contato no Fale Conosco e não deixe de assinar a nossa newsletter.
Referências:
- Centers for Disease Control and Prevention. Human coronavirus types. https://www.cdc.gov/coronavirus/types.html. Acessado em 17/03/2020.
- Jiang X., Rayner S., Luo M- Does SARS-CoV-2 has a longer incubation period than SARS and MERS? J Med Virol. 2020;92:476–478. https://doi.org/10.1002/jmv.25708. Acessado em 06/05/2020.
- Higuchi R., Dollinge, G., Walsh P. et al. Simultaneous Amplification and Detection of Specific DNA Sequences. Nat Biotechnol 10, 413–417 (1992). https://doi.org/10.1038/nbt0492-413. Acessado em 22/04/2020.
- CDC 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Diagnostic Panel. Instructions for use. Central for Disease Control. CDC-006-00019, Revision: 03 (2020). Acessado em 22/04/2020.
- Corman V. M. et al. “Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR.” Euro surveillance : bulletin Europeen sur les maladies transmissibles = Euro- pean communicable disease bulletin vol. 25,3 (2020): doi:10.2807/1560-7917.ES.2020.25.3.2000045. Acessado em em 22/04/2020.
- Wang W., Xu Y., Gao R., et al. Detection of SARS-CoV-2 in Different Types of Clinical Specimens. JAMA. Published online March 11, 2020. doi:10.1001/jama.2020.3786. Acessado em 22/04/2020.
